ロバスト線形回帰を扱うRの関数 rlm {MASS} について、引数 method を M推定 とした場合における、引数 scale.est の2つの選択肢、MAD(中央絶対偏差(Median Absolute Deviation)) と Huber それぞれの結果をシミュレーションで比較します。
本ポストは以下の資料を参照引用しています。
- https://www.stat.go.jp/training/2kenkyu/ihou/76/pdf/2-2-767.pdf
- https://www.st.nanzan-u.ac.jp/info/gr-thesis/ms/2004/kimura/01mm029.pdf
- https://stat.ethz.ch/R-manual/R-devel/library/MASS/html/lqs.html
前述の scale.est はロバスト線形回帰における 残差尺度(スケール) を指定する引数です。
M推定 では回帰係数 \(\beta\) を次の目的関数を最小化することで推定します。
\[\underset{\beta}{\textrm{min}}\displaystyle\sum_{i=1}^n\rho\left(\dfrac{y_i-x_i^{T}\beta}{\sigma}\right)\] ここで、\(\rho\) は損失関数、\(\sigma\) は残差の残差尺度パラメータ。
scale.est method of scale estimation: re-scaled MAD of the residuals (default) or Huber’s proposal 2 (which can be selected by either “Huber” or “proposal 2”).
出所:https://cran.r-project.org/web/packages/MASS/MASS.pdf
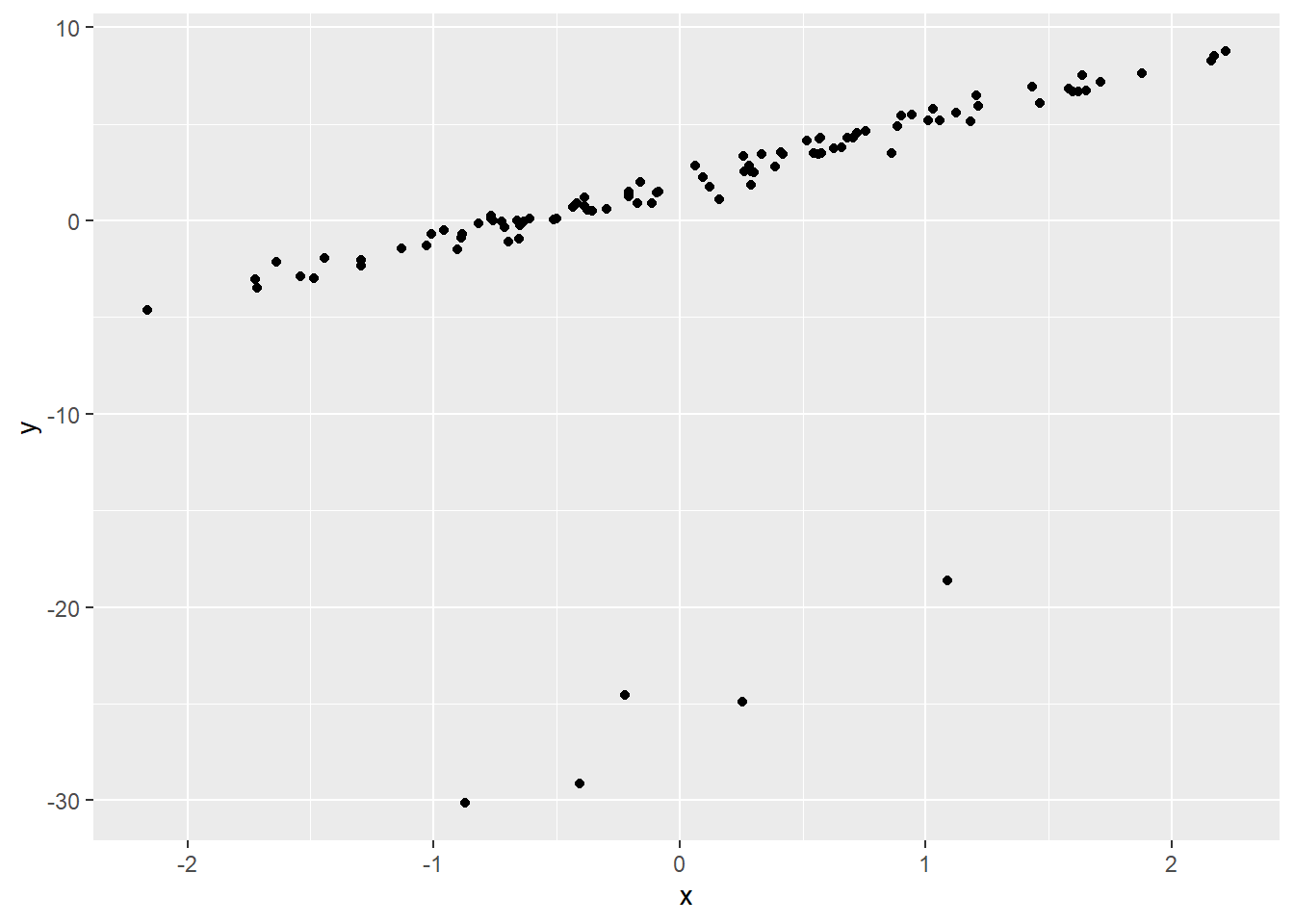
始めに外れ値のあるサンプルデータを生成する関数を作成します。
なお、外れ値は全て「意図的に大きな外れ」とし、かつ回帰直線を「下方向に引っ張る」値とします。
fun_sampledata <- function(seed, n, nout) {
set.seed(seed)
# 真のモデル: y = 2 + 3x + ε
x <- rnorm(n)
y_true <- 2 + 3 * x
y <- y_true + rnorm(n, sd = 0.5)
# 外れ値追加
id_out <- sample(x = seq(n), size = nout, replace = F)
y[id_out] <- y_true[id_out] + runif(n = nout, min = -30, max = -20)
data <- data.frame(x = x, y = y)
return(data)
}サンプルサイズは100点。うち5点(全体の5%)を外れ値としたサンプルです。
library(dplyr)
seed <- 20250325
n <- 100
nout <- 5 # 外れ値数
data <- fun_sampledata(seed = seed, n = n, nout = nout)
glimpse(data)Rows: 100
Columns: 2
$ x <dbl> 0.41244091, -0.29726921, 0.75426413, -1.02650381, -0.90249637, -0.37…
$ y <dbl> 3.51826312, 0.61542828, 4.63224788, -1.29074946, -1.50497772, 0.5556…外れ度合い をチャートで確認します。
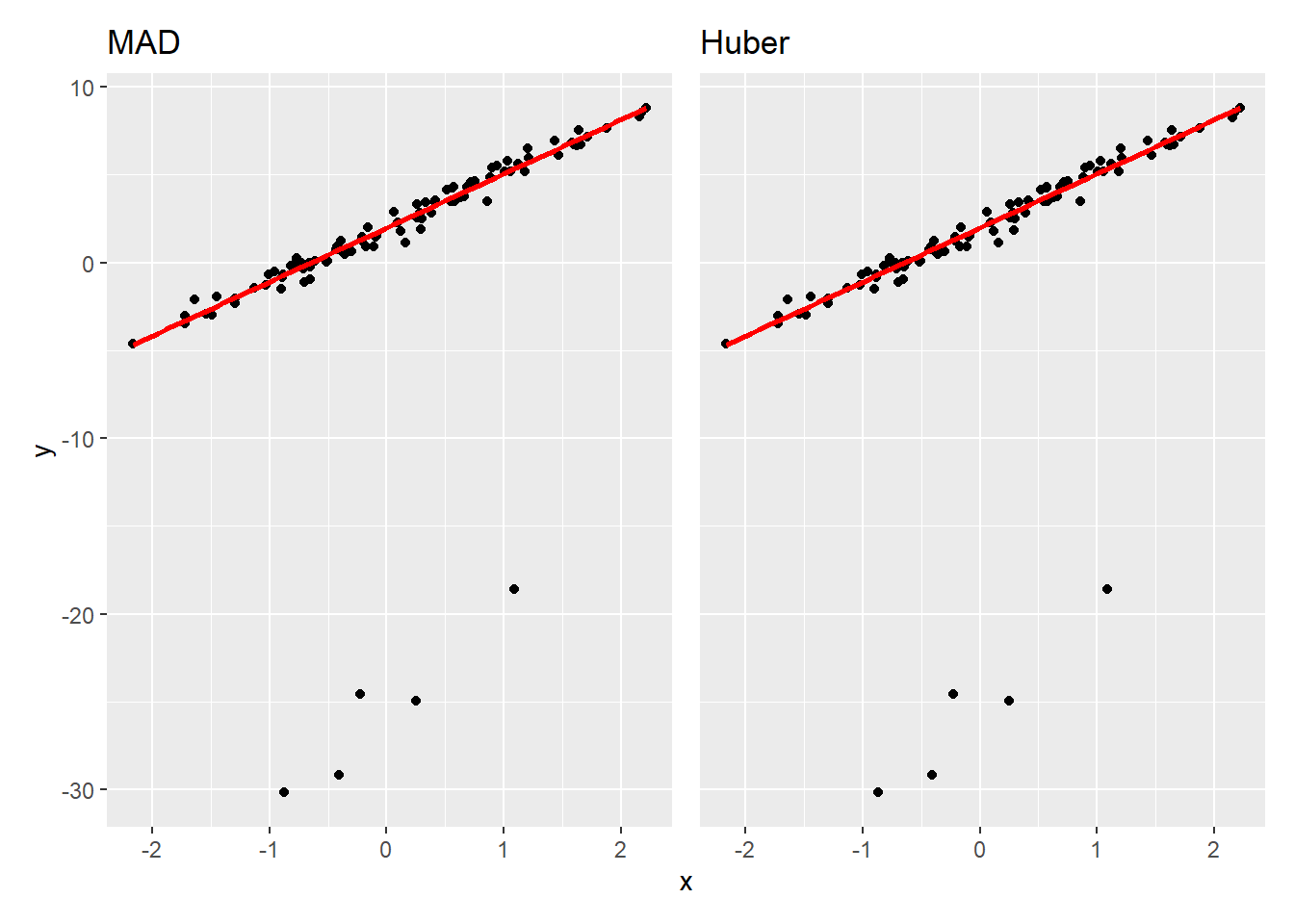
作成したサンプルデータについて、残差尺度 を MAD および Huber とした結果をチャートで比較します。
library(MASS)
library(patchwork)
g_MAD <- ggplot(data = data, mapping = aes(x = x, y = y)) +
geom_point() +
geom_smooth(method = "rlm", se = F, color = "red", method.args = list(method = "M", psi = psi.bisquare, scale.est = "MAD")) +
labs(title = "MAD")
g_Huber <- ggplot(data = data, mapping = aes(x = x, y = y)) +
geom_point() +
geom_smooth(method = "rlm", se = F, color = "red", method.args = list(method = "M", psi = psi.bisquare, scale.est = "Huber")) +
labs(title = "Huber")
g_MAD + g_Huber + plot_layout(axes = "collect")目視では MAD と Huber に差はほぼ見られませんが、
list(MAD = rlm(data$y ~ data$x, method = "M", psi = psi.bisquare, scale.est = "MAD"), Huber = rlm(data$y ~ data$x, method = "M", psi = psi.bisquare, scale.est = "Huber"))$MAD
Call:
rlm(formula = data$y ~ data$x, psi = psi.bisquare, scale.est = "MAD",
method = "M")
Converged in 4 iterations
Coefficients:
(Intercept) data$x
1.974910 3.075421
Degrees of freedom: 100 total; 98 residual
Scale estimate: 0.484
$Huber
Call:
rlm(formula = data$y ~ data$x, psi = psi.bisquare, scale.est = "Huber",
method = "M")
Converged in 7 iterations
Coefficients:
(Intercept) data$x
1.975536 3.075422
Degrees of freedom: 100 total; 98 residual
Scale estimate: 0.471 実際には 切片、傾き(僅か)、そして 残差尺度 全てに差が生じています
それでは300試行のシミュレーションにより、残差尺度を MAD および Huber とした場合それぞれのロバスト線形回帰の係数(切片 と 傾き)と 残差尺度 を比較します。
fun_simulation <- function(iter, n, nout) {
formulatxt <- sampledf$y~sampledf$x
method <- "M"
psi <- psi.bisquare
maxit <- 100
for (iii in seq(iter)) {
sampledf <- fun_sampledata(seed = iii, n = n, nout = nout)
# 係数の抽出
result.rlm.m.mad <-
rlm(formulatxt,
scale.est = "MAD",
method = method,
psi = psi,
maxit = maxit
)$coef
result.rlm.m.huber <-
rlm(formulatxt,
scale.est = "Huber",
method = method,
psi = psi,
maxit = maxit
)$coef
resultdf0 <- rbind(result.rlm.m.mad, result.rlm.m.huber) %>%
t() %>%
data.frame(check.names = F) %>%
{
.$itr <- iii
.
}
if (iii == 1) {
resultdf_coef <- resultdf0
} else {
resultdf_coef <- rbind(resultdf_coef, resultdf0)
}
# スケールの抽出
result.rlm.m.mad <-
rlm(formulatxt,
scale.est = "MAD",
method = method,
psi = psi,
maxit = maxit
)$s
result.rlm.m.huber <-
rlm(formulatxt,
scale.est = "Huber",
method = method,
psi = psi,
maxit = maxit
)$s
resultdf0 <- data.frame(result.rlm.m.mad, result.rlm.m.huber, itr = iii)
if (iii == 1) {
resultdf_scale <- resultdf0
} else {
resultdf_scale <- rbind(resultdf_scale, resultdf0)
}
}
# 切片
tidydf <- resultdf_coef %>%
row.names() %>%
grep("Intercept", .) %>%
resultdf_coef[., ] %>%
{
tidyr::gather(., , , colnames(.)[-3])
}
g_coef_intercept <- tidydf %>% ggplot(mapping = aes(x = "", y = value)) +
geom_boxplot() +
geom_jitter() +
facet_wrap(. ~ key, scales = "free_y") +
theme(axis.title = element_blank()) +
labs(title = "Intercept")
# 傾き
tidydf <- resultdf_coef %>%
row.names() %>%
grep("sampledf", .) %>%
resultdf_coef[., ] %>%
{
tidyr::gather(., , , colnames(.)[-3])
}
g_coef_slope <- tidydf %>% ggplot(mapping = aes(x = "", y = value)) +
geom_boxplot() +
geom_jitter() +
facet_wrap(. ~ key, scales = "free_y") +
theme(axis.title = element_blank()) +
labs(title = "Slope")
# スケール
tidydf <- resultdf_scale %>%
{
tidyr::gather(., , , colnames(.)[-3])
}
g_scale <- tidydf %>% ggplot(mapping = aes(x = "", y = value)) +
geom_boxplot() +
geom_jitter() +
facet_wrap(. ~ key, scales = "free_y") +
theme(axis.title = element_blank()) +
labs(title = "Scale")
g_coef_intercept + g_coef_slope + g_scale + plot_layout(nrow = 3)
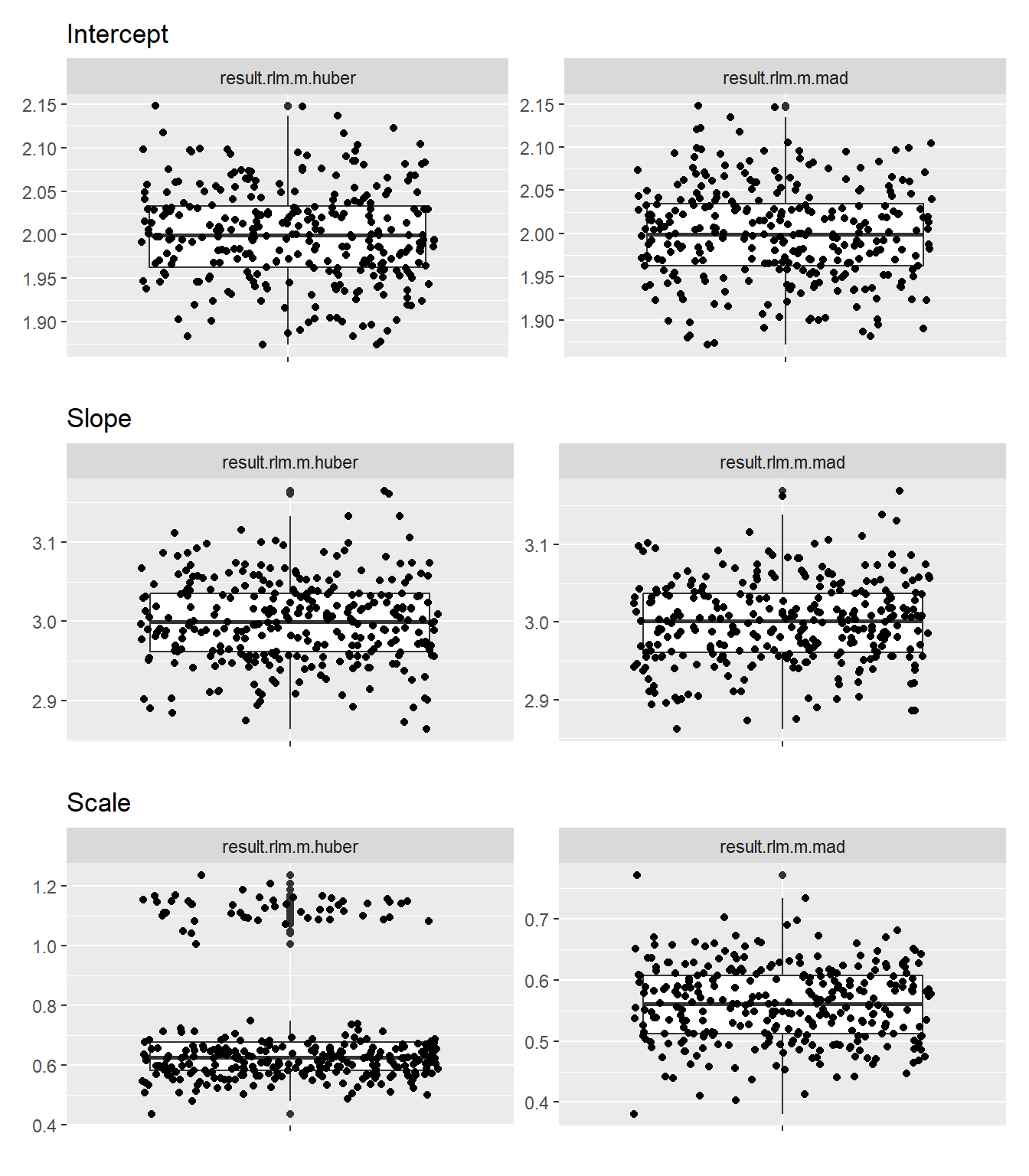
}始めに外れ値を全体の10%とした場合を比較します。
残差尺度 には違いが見られますが、切片 と 傾き は中央値および分散とも MAD と Huber とで、ほぼ同じ結果になりました。
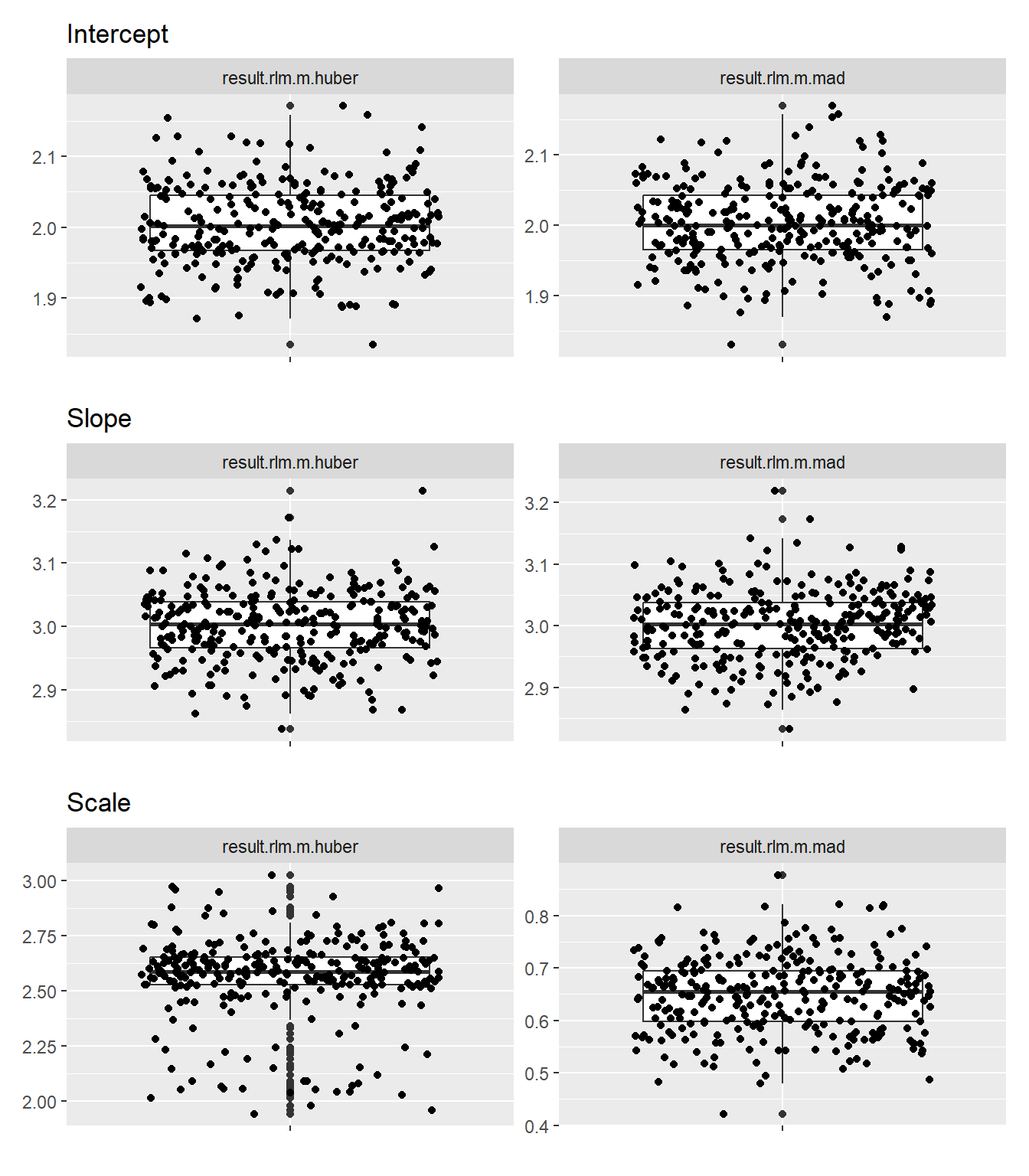
続いて外れ値を全体の30%にした場合です。
残差尺度 の違いはより大きくなりましたが、それでも 切片 と 傾き は中央値および分散とも MAD と Huber とで、ほぼ同じ結果になりました。
以上です。